고정 헤더 영역
상세 컨텐츠
본문
작성자 : 채윤병
참고 : MIT spring 2021 6.874 Computational system biology lecture 15
일반적인 ML, deep learning의 경우 데이터의 형태가 이미지, 텍스트, 수치와 같은 형태
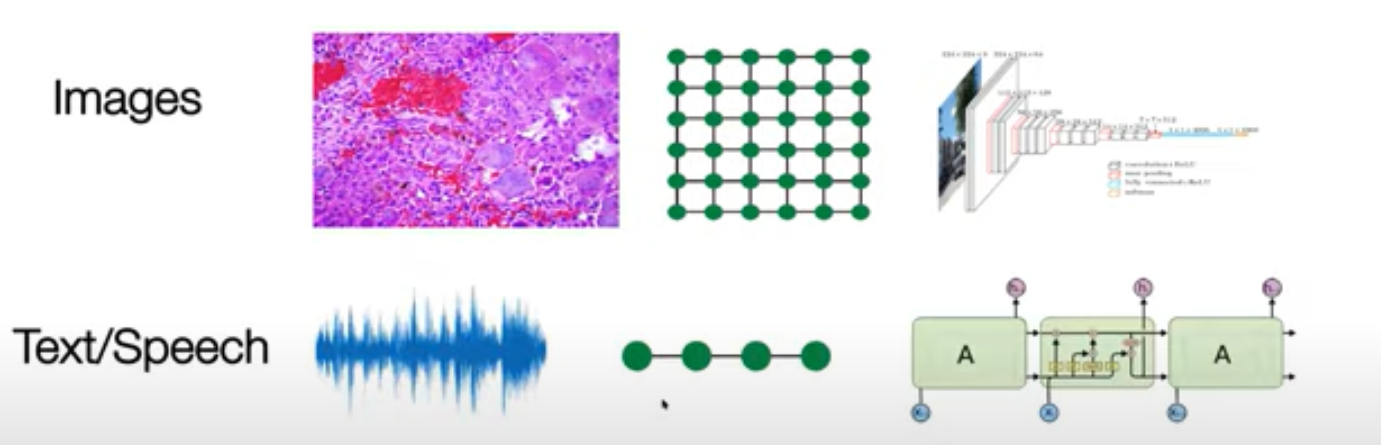
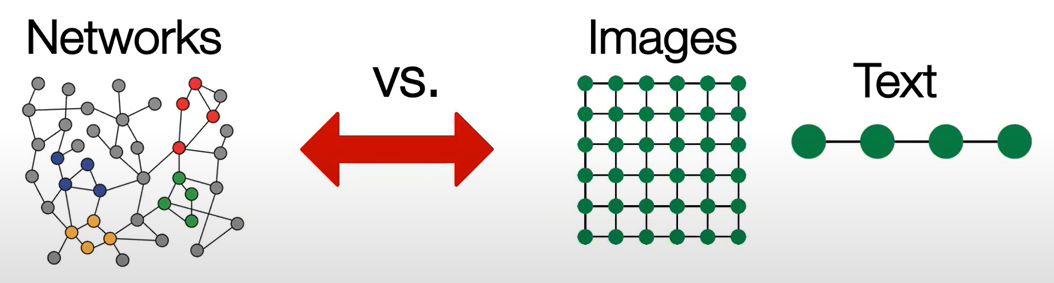
생물 데이터(gene-gene, cell-cell, protein-protein)의 경우 데이터간의 연관성이 매우 복잡하게 얽혀있기 때문에 일반적인 데이터 형태로 표현하기가 어려움 >> Graph network 형태로 데이터를 표현
데이터의 형태에서 Node는 drug discovery의 경우 약물이 될 수도 있고 gene, protein등이 될 수 있다. Edge의 경우 Node들 간의 연관성을 담고 있다.
(그렇지만 Drug discovery와 같은 분야에서 일반적인 ML모델 또한 광범위하게 사용되고 있다. 측정 기술의 발달로 예전보다 세밀한 범위(예전에는 조직 단위 > 현재는 세포하나의 단위)에서의 측정이 가능해졌기 때문에 얻을 수 있는 데이터의 양이 방대해졌다. 이렇게 얻은 데이터를 가지고 머신러닝은 사람이 판단할 수 없는 부분까지 편향을 가지지 않고 예측을 할 수 있기때문에 Drug discovery에서 머신러닝이 중요한 역할을 하고 있다.)
Graph 형태의 data 특징 :
1. Arbitrary size : Node의 수, interaction(edge)의 수에 따라서 데이터의 크기가 변화
2. Node간의 순서나 공간적인 정보가 없음
네트워크를 분석하기 위해서는? > 네트워크를 분석가능한 형태로 만들어야 함 (노드의 feature, adjacency matrix, vertex set, etc.)
Node feature로는 gene expression profile, functional information등이 될 수 있음.
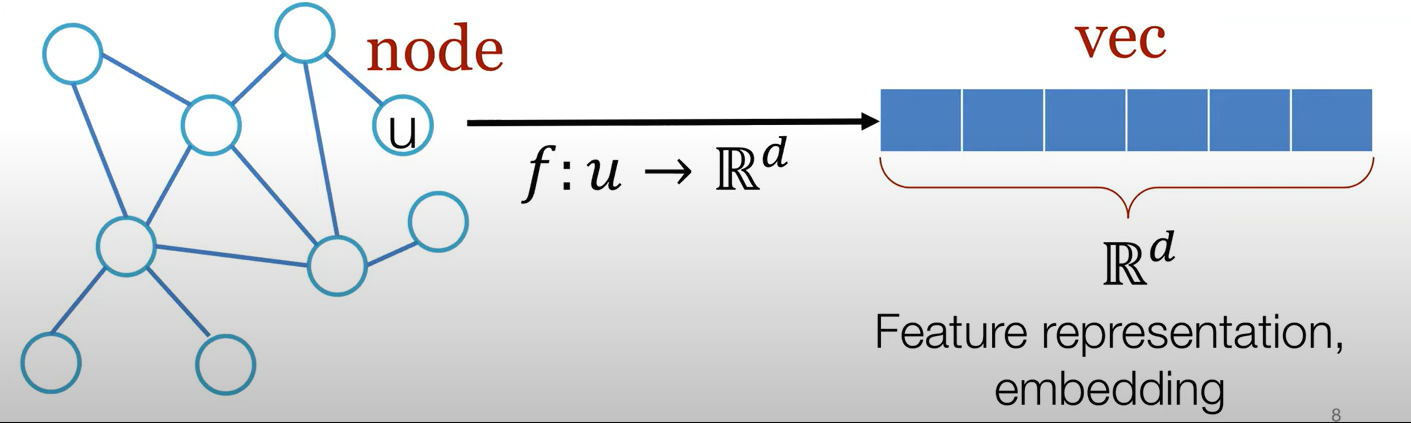
목적 : 그래프 형태의 데이터를 학습(분석)이 가능하도록 embedding을 해주는 것
V : Vertex set (노드의 집합)
A : Adjacency matrix, 인접 행렬 (노드간 연결관계를 담고 있는 행렬)
X : R^(mxV)의 matrix (노드의 특성, m은 특성의 개수, V는 노드 집합)
N(v) : 이웃 노드의 개수
1. A Naive approach
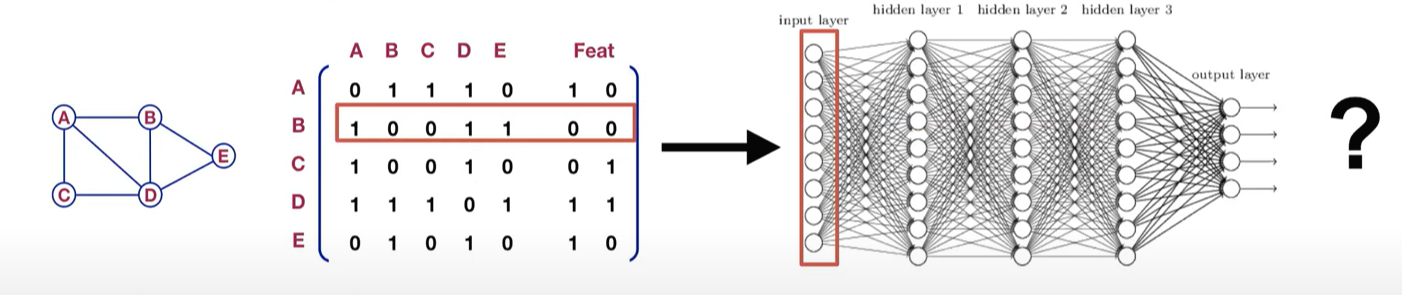
문제점 :
- 파라미터가 너무 많음
- 그래프의 크기에 따라 embedding이 되기 때문에 다른 크기의 그래프에는 사용할 수 없음
- Node의 순서에 따라 결과가 달라짐
2. Graph convolution

그래프 형태의 데이터를 효과적으로 학습시킬 수 있는 방법. 어떻게 Graph convolution으로 embedding을 할 수 있을까?
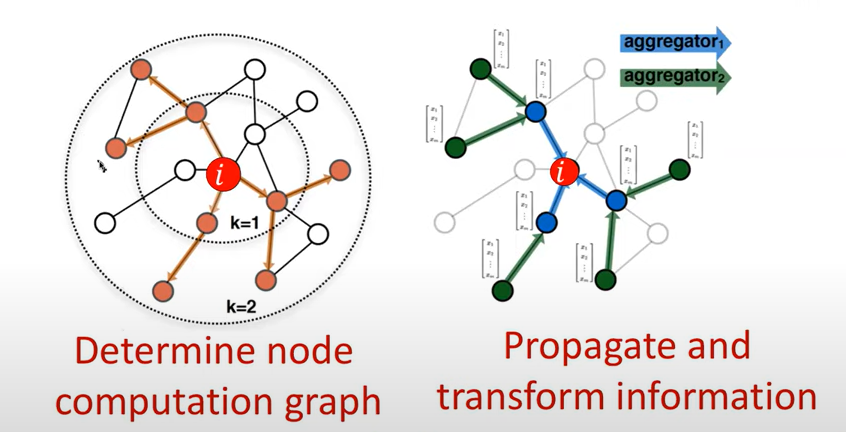
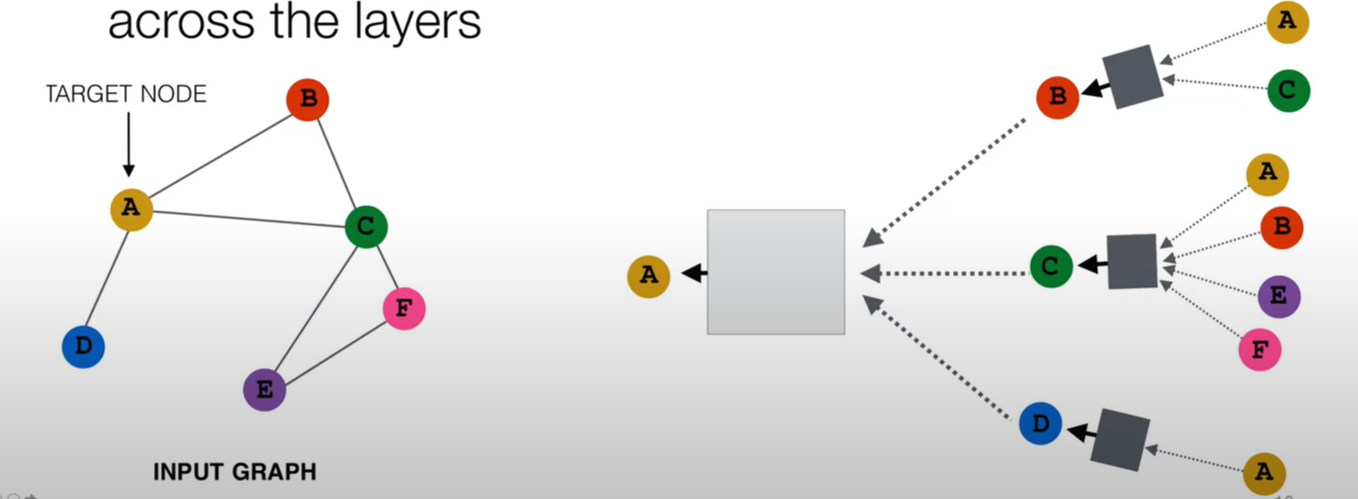
Aggregation 과정의 진행 :
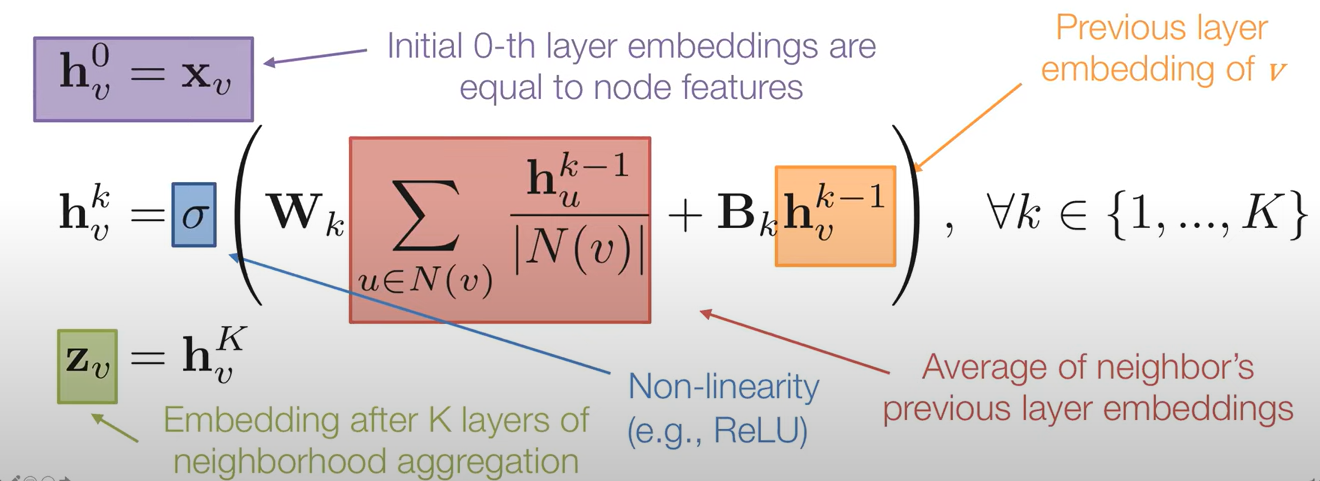
2-1 Basic approach : 이웃 노드로부터 정보들의 평균을 구하고 이를 신경망에 적용
2-2 GraphSAGE
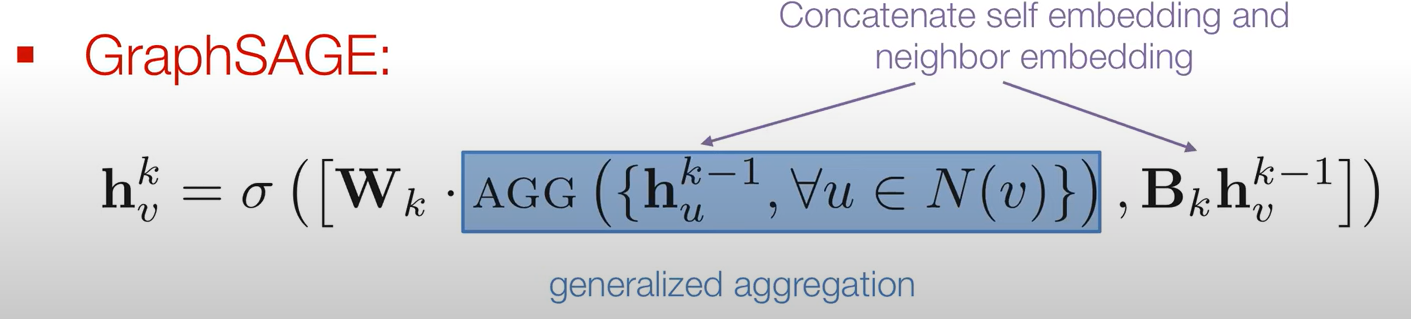
Basic approach에서는 단순히 평균을 냈다면, GraphSAGE에서는 pooling을 적용하는 방식, lstm을 적용하는 방식 등 더욱 다양한 방법이 있다. 구체적인 설명은 나와있지 않다..!
GCN(Graph convolution network)을 이용한 결과

2개의 convolutional network를 사용하였고 A hat은 normalized된 Adjacency matrix, 마지막 layer에 softmax를 통과시켜 노드의 classification을 진행한 결과 다른 모델보다 GCN의 성능이 더 좋았다!
2개의 layer만 사용한 이유? > layer를 많이 넣게되면 over smoothing이 되는 문제가 있어서 2개의 layer만 사용했다고 함.
Drug discovery에서의 Graph network 활용

Drug discovery에서는 node와 edge의 유형을 구별할 필요가 있다.
Node의 경우 약물, 단백질, 유전자 등 다른 종류의 node들이 존재하고 edge의 경우 약물과 약물간의 상호작용은 단백질과 단백질 사이의 상호작용과 다르고 약물과 단백질 사이의 상호작용과도 다르기 때문에 edge의 유형별로 나누어서 embedding을 진행한다.
Vt : vertex set (t유형 노드의 집합)
Ar : adjacency matrix (유형 r(edge)의 노드간 연결관계를 담고 있는 행렬)
Xt : R^(mxV)의 matrix (t유형 노드의 특성, m은 특성의 개수, V는 노드 집합)
> drug discovery의 경우 노드로는 단백질, 유전자, 약물이 될 수 있어서 유형을 나누는 것이 필요하며 노드간의 관계 또한 유형별로 나누어야한다.



네트워크의 분석으로 알 수 있는 정보 :
- Node의 유형 예측 (Node classification) -> softmax(zn)
- 두 노드의 연결 예측 (Link prediction) p(Aij) -> sigmoid(zi.T*zj)
- 노드의 clustering (Community detection)
- 두 네트워크의 유사도 (Network similarity)
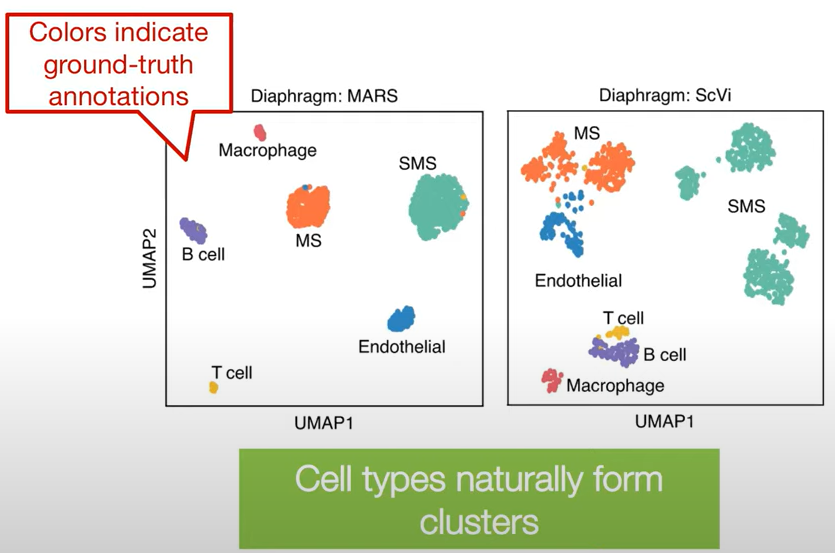
+ Meta learning - 미리 학습된 정보들로 embedding 되지 않은 cell type을 embedding 할 수 있음!
MARS - meta learning method
Annotated experiment data로 이루어진 meta-dataset으로 unannotated experiment data를 optimize하고 이를 이용해 다시 meta-dataset도 optimize하는 방식
감사합니다!
'심화 스터디 > Graph Study' 카테고리의 다른 글
| [Graph 스터디] Semi-supervised classification with Graph Convolution Networks (0) | 2022.11.10 |
|---|---|
| [Graph 스터디] Neural Message Passing for Quantum Chemistry(MPNN) (0) | 2022.11.02 |
| Predict then propagate : Graph Neural Network meet Personalized PageRank (0) | 2022.09.22 |
| Graph neural network review(2) (0) | 2022.09.13 |
| Graph neural network review(1) (1) | 2022.09.13 |





댓글 영역