고정 헤더 영역
상세 컨텐츠
본문 제목
[CV 논문 리뷰 스터디 / 2주차 / 장수혁] U-Net: Convolutional Networks for Biomedical Image Segmentation, 2015
본문
Abstract
기존까지 deep network들을 성공적으로 훈련시키려면 필연적으로 수천개 이상의 annotated된 training sample이 필요했음.
본 논문에서 제시하는 네트워크와 training 방법은 data augmentation에 focus를 하여 available한 annotated sample을 더 효과적으로 활용할 수 있게 하였음.
U-Net은 context를 포착하는 contracting path와 정확한 localization을 위한 expanding path로 구성되어 있고, 이 두 path는 서로 symmetric.(localization = 이미지 안에서 bbox등을 사용하여 객체의 위치 정보를 출력해 주는 것(bbox에선 왼쪽 위, 오른쪽 아래 좌표 출력))
U-Net은 매우 적은 이미지를 가지고 end-to-end로 학습하여도 기존의 best method인 sliding-window cnn방식을 outperform하였다.(on the ISBL challange)
U-Net은 512*512 image를 segmentation할 때 최신 GPU상에서 1초가 안걸릴정도로 처리속도도 빠르다.
Introduction
CNN을 활용한 모델들은 오랫동안 visual recognition부문에서 SOTA를 차지했었지만, training set과 network의 size에 제약을 받는 다는 한계가 있었다. 100만개의 training image가 있는 ImageNet dataset을 활용한 8개의 레이어를 가진 큰 네트워크가 나오면서부터 breakthrough가 생기기 시작했으며, 이후 더 크고 깊은 network들이 출현하게 되었다.
CNN 모델들은 주로 image당 single class label을 할당하는 classification task에 적용되었으나, biomedical image processing을 포함한 많은 visual task들에선 output이 localization을 포함해야만 하였다. 즉, class label이 each pixel이 할당되어야 했고 이것이 바로 image segmentation task라고 할 수 있다.
이런 이유로, sliding-window 기법을 활용한 network가 patch(local region)를 활용하여 each pixel의 class label을 예측하려고 하였다. 이 network는 1.localize가 가능했으며 2.training image의 수보다 patch의 수가 더 많았다는 특징이 있었다.
하지만 이 방법에는 2가지 결점이 있었다.
1. Quite slow
- network가 each patch마다 separately하게 실행되어야 했다.
- overlapping patch들로 인해 redundancy가 많이 발생하였다.
2. trade-off between localization accuracy and the use of context
- patch가 크면 넓은 범위의 이미지를 인식하는데 뛰어나 context 인식 탁월, but more max-pooling layer로 인해 localization accuracy 떨어짐
- patch가 작으면 localization은 뛰어나나, only little context를 볼 수밖에 없음
본 논문에서는 소위 'fully convolutional network"라 할 수 있는 elegant한 architecture를 소개한다.
U-Net은 1. 매우 적은 이미지로 2. 더 정확한 segmentation을 수행할 수 있다.

U-Net은 contracting network에서 축소된 이미지를 연속적인 layers(upsampling operator)로 보충하여 다시 output의 해상도를 높여가는 구조로 이루어져 있다. 즉, localize를 하기 위해 contracting path에서 나온 high resolution features들이 upsampled output과 결합되는 것이다.
여기서 주목할 점은, upsampling part(expanding path)에서도 large number of feature channels를 갖는다는점이다. 이것은 contracting path에서 추출한 context information을 high resolution layers로 전파하는데 유용하다. 결과적으로, contracting path와 expanding path는 거의 symmetric하고 U-shape을 띄게 된다.
U-Net은 fully connected layers를 사용하지 않고 각 convolution의 valid part만 사용한다. 예를 들면, segmentation map은 full context가 input image에 존재하는 pixels들만 포함하고 있다. 이것은 overlap-tile strategy에 의한 매끄럽고 연속적인 segmentation을 가능하게 한다.
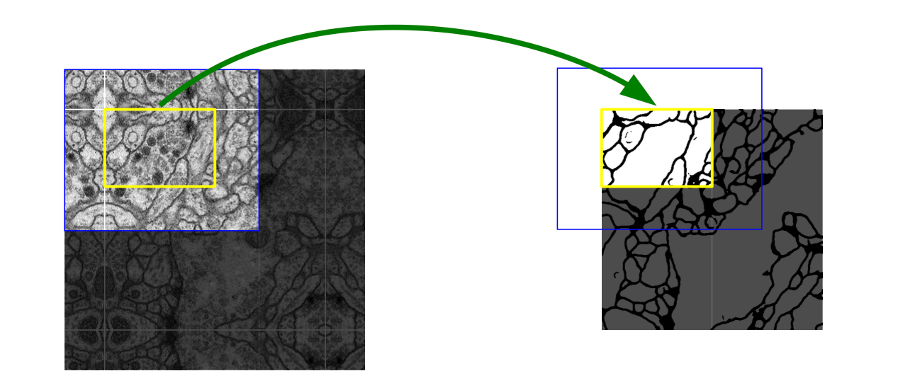
위 이미지는 Overlap-tile strategy를 설명하는 이미지인데, 파란색 영역이 input이고 노란색 영역이 그 input에 대한 segmentation 예측값이다. 노란색 영역 밖의 파란색 영역은 missing input data로, 이 영역은 mirroring을 통해 추론한다.(노란색 영역의 경계에 거울을 갖다댄 것처럼 대칭시킨다) 이 방식은 GPU memory 때문에 해상도가 제한받는 것을 없애주므로, large image에 U-Net을 적용하는데 아주 유용하게 쓰인다.
본 논문에서는 task를 해결하기 위해 아주 작은 양의 training data만 사용했기에, 필연적으로 data augmentation을 사용하였고 특히 elastic deformation을 적용하였다. 이것은 network가 deformations에 대해 invariance를 유지하게끔 하였다. 특히 biomedical segmentation의 경우, 세포나 조직에 deformation이 가해지는 것이 가장 흔한 variation이고 실제와 유사한 deformation이 효과적으로 simulated되기 때문에 더더욱 중요하다.
또 다른 segmentation task challenge에는 the separation of touching objects of the same class가 있다. 이를 해결하기 위해 저자들은 weighted loss를 사용하는 것을 제안하였고, 이것은 touching cell들 사이에 있는 separating background labels에 대해 큰 weight를 부여한다.
결과적으로 U-Net은 다양한 biomedical segmentation problem에 적용할 수 있었으며, 여러 데이터셋에서 좋은 성과를 보여주었다.
U-Net's Network Architecture
- contracting path(left side)와 expansive path(right side)로 이루어져있다.
- contracting path는 double 3*3 conv(unpadded) > ReLU > 2*2 maxpooling(stride 2 for downsampling) 구조로 이루어져 있다. downsampling step에서 feature channel들은 2배가 된다.
- expansive path에서는 먼저 2*2 up convolution을 바탕으로 한 upsampling feature map과 contracting path에서 cropped 된 feature map들이 concat(추후의 encoding-decoding 방법과 유사)된다.(conv과정에서 경계 pixel들에서 loss가 발생하기 때문에 crop이 필수) upsampling step에서 feature channel들은 절반으로 감소한다. 이후 contracting path와 유사하게 double 3*3 conv > ReLU 가 따라온다.
- expansive path에서 마지막으로 1*1 conv layer가 나오게 되는데, 기존 layer의 64개 channel을 2개의 channel로 바꾸고 이미지 크기는 유지하면서 64개의 feature vector를 원하는 class에 할당하는 역할을 한다.
- 최종적으로 U-Net은 23개의 convolutional layers를 가지고 있다.
- seamless한 output segmentation map을 만들기 위해서 input tile size를 contracting path에서 모든 2*2 maxpoolng이 원활하게 적용될 수 있게 끔 짝수로 맞춰주는 게 좋다.(even x- and y-size)
Training
- U-Net은 SGD(stochastic gradient descent)방식을 이용해 학습된다.
- unpadded convolution 연산을 진행하기 때문에 필연적으로 output image가 input image보다 일정한 너비만큼 작을 수밖에 없다.
- GPU를 최대로 사용하기 위해 batch size는 작게 가져가는 대신 intput tile의 size를 키우는 것을 택했다. 또한, momentum값을 0.99로 매우 크게 가져가서 기존의 학습방향을 많이 따라가는 방식을 취했다.(batch size를 줄인 것에 대한 보완)
- Energy function은 cross entropy와 final feature map에서의 pixel-wise soft-max를 결합하여 계산된다.

위 수식은 pixel-wise soft-max를 의미하며 ak(x)는 pixel position x에서 feature channel k에 대한 activation으로 확률값이라고 생각하면 된다. 그렇게 해서 모든 클래스에 대하여(K: 클래스의 개수) soft-max를 진행한다. 만약 pk(x)가 1에 가깝다면, 그때의 ak(x)는 maximum activation이고 나머지는 모두 0에 가까울 것이다.

위의 energy function은 pl(x)(x)의 편차에 대한 penalty를 준 cross entropy와 weight map이 결합된 형태이다. 여기서 l(x)는 각 픽셀의 실제 true label이고, 로그 특성상 각 픽셀의 정답 라벨에 대한 activation이 작게 되면 큰 페널티를 받게 된다. weight map은 이전에 언급했던 touching cell들 사이에 있는 border pixel들에 대해 더 중요성을 부여하기 위해 설계되었다. 또한 각 정답 segmentation의weight map은 특정 class에만 편재되어있는 pixel의 frequency를 보정하기 위해, 작은 구분 경계들을 학습하기 위해 사전 계산된다.

weight map은 class frequencies를 조절하는 wc(x)와 가장 가까운 셀과 두번째로 가까운 셀의 경계까지에 거리에 따른 가중치 부분으로 나누어져 있다. 셀간의 거리가 가까우면 가까울수록 weight map의 값은 구분을 위한 본래의 목적대로 커지게 되어있다.
- 이러한 가중치의 적절한 초기화는 매우 중요한 포인트이다. 초기화가 잘 이루어지지 않으면 특정 파트에 과한 activation이 부여되고, 다른 부분은 아예 activation이 일어나지 않을 수 있다. U-Net에서는 가우시안 분포를 활용하여 가중치를 초기화하였다.
Data Augmentation
Data augmentation은 training data가 소량 밖에 없을 때, network를 invariant하고 robust하게 학습시키기 위해 필수적인 과정이다.
현미경 이미지에 있어서 저자들은 deformation에 invariant하고 robust하기 위해 shift와 rotation 기법을 주로 사용했었다. 특히 본 논문에서는 U-Net을 소량의 annotated image로 학습시키는데 random elastic deformation이 key augmentation 역할을 하였다.
Conclusion
U-Net은 여러 biomedical segmentation applications에 대해 매우 좋은 성능을 달성하였다. Elastic deformation data augmentation을 활용한 덕분에, 매우 적은 annotated image을 가지고 매우 합리적인 training 시간(6GB GPU로 10시간)만에 좋은 모델을 구축할 수 있었다.
Reference





댓글 영역