고정 헤더 영역
상세 컨텐츠
본문 제목
[CV 논문 리뷰 스터디 / 2주차 / 최경석] U-Net: Convolutional Networks for Biomedical Image Segmentation(MICCAI 2015)
본문
논문 : https://arxiv.org/abs/1505.04597
U-Net: Convolutional Networks for Biomedical Image Segmentation
There is large consent that successful training of deep networks requires many thousand annotated training samples. In this paper, we present a network and training strategy that relies on the strong use of data augmentation to use the available annotated
arxiv.org
0. Segmentation이란?
Image Segmentation : 이미지를 픽셀별로 분할하는것 / 이미지의 모든 픽셀에 대해 라벨을 할당하는 task

0.1. Semantic Segmentation
Semantic Segmentation은 같은 class이면 같은 영역으로 인식한다.
각 픽셀별로 어떤 class에 속하는지 labeling(Classification)하는 것이 목표


Class의 개수만큼 출력 채널(H×W×N)을 만든 후에 pixel들이 각 class에 대해 포함되는지 판단한 이후, one-hot encoding으로 binary한 값을 가지도록 한다.
첫번째 layer에서는 해당 픽셀이 사람에 포함되는 픽셀인지 등등등,,,,
0.2. Instance Segmentation
Instance Segmentation은 같은 class여도 서로 다른 object에 대해서는 각각 구분한다.
각 픽셀별로 object가 있는지 없는지 여부를 판단하는 것이 목표
Bounding Box를 통해서 object를 먼저 localizing 시켜준 이후에, 그 ROI안에서 class 개수 만큼 masking한다.

위 그림은 Mask R-CNN의 architecture인데, 저번에 했던 object detection의 일종인 Faster R-CNN을 baseline으로 사용한다.
Faster R-CNN을 통해 얻은 ROI에 대해 기존의 classification & bbox regression Task 와 동시에 Segmentaiton을 진행하는 mask branch를 추가한 구조이다.
Mask R-CNN의 경우에는 각각의 ROI에 FCN(Semantic Segmentaiton)을 사용하여 Segmentation 진행하였다.
Faster R-CNN은 spatial 정보를 유지하기 위해 ROI Pooling 대신 ROI Align 기법을 사용하였는데, 내용은 다음과 같다.
* 저번학기에 진행한 Image Study에서 했던 자료 참조



0.3. Panoptic Segmentation
Panoptic Segmentation은 Semantic Segmentation과 Instance segmentation을 결합한 방식이다.
각 Object를 개별 Instance로 구분함과 동시에, Class를 예측하는 것이 목표
Panoptic Segmentation은 셀 수 있는 Class(사람, 자동차,,)과 셀 수 없는 Class(하늘, 도로, 땅,,,)를 구하는데,
각각 Thing, Stuff라고 한다.
Thing Class에 대해서는 Instance Segmentation
Stuff Class에 대해서는 Semantic Segmentation
을 진행한다.
1. Introduction
전통적으로 CNN은 한개의 label을 예측하는 image classification task에서 좋은 성능을 보였다.
그러나 여러 computer vision task에서는, 특히 biomedical 분야에서는 localization taks의 역할 또한 매우 중요하다. 즉, 세포 이미지 같은 경우에서도 미세하게, pixel별로 어떤 class label에 속하는지 판단하는 Semantic Segmentation 기법이 사용되어야한다.
또한, biomedical 분야에서는 기존에 imagenet 데이터셋과 같이 많은 양의 training image를 구할 수도 없다!!
기존에 사용하던 sliding-window 기법을 통한 Semantic Segmentation에는 2가지 큰 문제점이 있었다.

1) 각 sliding window에 대해 각각 network를 거쳐야해서 속도가 느렸고, 겹치는 patch로 인해서 중복성이 크다.
2) Localizing accuracy와 use of context 사이에 trade-off가 존재한다. 즉, Patch가 크면 localizing이 잘 안되고, 작으면 주변 context를 잘 못본다
논문에서는 FCN 기반으로 적은 image로 높은 성능을 낼 수 있는 새로운 architecture을 개발하였고, 핵심적인 내용은 다음과 같다. (Skip Connection 기법)
1. Contracting Path + Expansive Path (Contracting Path의 Feature를 Copy&Crop)
2. 효율적인 Data Augmentation
3. Seperation of touching object - Weighted Loss
내용이 많지만 뒤에서 차차 한개씩 설명할 예정이다.
2. Network Architecture
U-Net 구조의 핵심적인 아이디어는 Contracting Network 뒤에 Pooling Operator 대신 Up-Sampling Operator을 추가한 것이다.
Contracting Network는 일반적인 convolutional network의 형태를 가지고 있다.
더욱 높은 localizing 성능을 가지기 위해 Contracting Network를 통해 얻어진 고차원 feature map과 Up-Sample된 output이 합쳐지도록 구성하였다.

2.1. Down Sampling & Up-Sampling

Down Sampling : 일반적인 Convolution 방식으로, Feature Map의 크기를 작아지게 한다.
Up Sampling : Down Sampling과 반대로, Feature Map의 크기를 커지게 하고, 해상도를 증가시킨다.
2.2.. Contracting Path
- Input : 572×572×1
- 2×2 Conv Filter
- Padding을 하지 않았기 때문에 Feature map의 크기가 2씩 감소
- Channel은 2배 증가
- 2×2 Max Pooling : 해상도(Feature Map의 H, W)가 2배 감소
- 일반 CNN처럼 <Conv -BN - ReLU - Conv - BN - ReLU - MaxPool> 반복
- Output : 32×32×512
해상도는 안좋지만, Context 및 개체 정보를 잘 포함 (Semantic)

2.3. Expansive Path
- 2×2 Conv Filter (Up-convolution)
- 해상도(Feature Map의 H, W)가 2배 증가
- Channel은 2배 감소
- Contracting Path에서 나온 Feature Map을 Copy&Crop한 후, 같은 레벨의 Expansive Path의 Feature Map에 Concat
- <UpConv - Concat - Conv - BN - ReLU - Conv - BN - ReLU> 반복
- 마지막에 1×1 Conv를 사용해서 2개의 class로 분류하는 segmentation map 생성
높은 해상도의 Segmentation 결과 산출 + Dense Prediction (정교한 Sementation)

Expansive Path에서 이전의 Contracting Path의 feature map을 사용함으로써 저차원(고해상도) 정보와 고차원(저해상도) 정보를 동시에 사용할 수 있다.
특히, Upsampling 과정(해상도 복원)에서도 많은 channel수를 유지하여 context 정보를 잘 유지할 수 있었다.
이러한 U 모양의 구조는 FC layer(fully connected layer)이 없고, convolution의 결과만을 사용한다. 또한 input image에 대한 전체 context가 사용 가능한 형태이다.
이러한 특징으로 임의의 image에 대해 "Overlap-Tile"기법을 사용하여 seamless segmentation이 가능하다.
2.4. Overlap Tile Strategy
Overlap Tile Strategy는 GPU에 영향을 받지 않고 높은 image resolution을 유지하기 위해 사용하는 기법이다.
Biomedical image는 이미지 크기가 매우 크기 때문에 보통 patch단위로 잘라서 Input으로 사용한다.
이를 "Tile"이라고 한다.
그리고, Unet에서는 padding 없이 architecture을 구성했기 때문에 이미지 크기가 계속 줄어들게 된다.
그렇기 때문에 애초에 더 큰 영역의 이미지를 사용해서 영역을 segmentation한다.

위의 예시처럼 왼쪽의 노란색 영역을 출력하기 위해 오른쪽의 파란색 영역 데이터를 사용한다.
가장자리 부분에 대해서는 이미지를 mirroring해서 사용했다. (Extrapolation)
Mirroring 한 이유는, 세포 같은 Biomedical image에서는 대칭 형태를 가진 경우가 많기 때문이다.
3. Training
U-Net은 SGD(Stochastic Gradient Descent)를 사용한다.
사용되는 Conv filter는 padding이 사용되지 않아 output image의 size가 더 작다.
Overhead(간접 처리 시간/메모리)를 줄이고 GPU 자원을 최대로 사용하기 위해 큰 batch size를 사용하는 것 보다, 큰 input tile을 사용하는 것을 선호한다.
또한, batch size가 작기 때문에 SGD의 momentum을 0.99로 크게 주어, 이전의 gradient descent값을 더 많이 반영하도록 하였다.
3.0. Loss Function
U-Net에서는 pixel-wise softmax function를 적용한 cross entropy loss function를 사용했다.


한편, Biomedical 영역에서 Segmentation에서는 동일한 클래스의 접촉 개체를 분리하는 것이 중요하다.
(세포 사진같은 경우에는 가까이 위치한 경우가 많기 때문에 명확히 구분하는 것이 중요)
위에서 언급했던 것처럼, 이걸 해결하기 위해 object 사이의 경계에 대해 weight(가중치)를 적용했다.
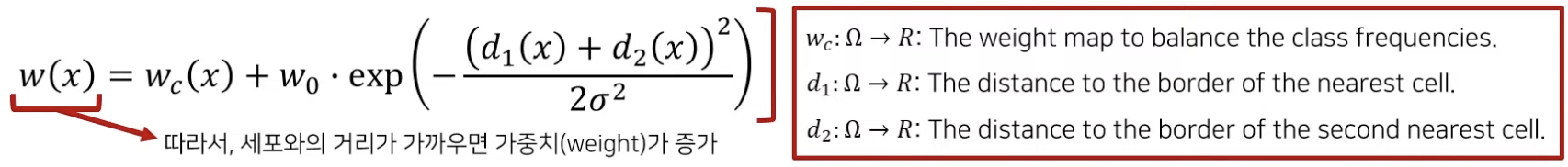
Wc : Class의 빈도에 따른 조정 값
d1(x) : 가장 가까운 세포의 경계와의 거리
d2(x) : 두번째로 가까운 세포의 경계와의 거리
이를 통해 인접한 셀 "사이에" 있는 배경 레이블에 대해 높은 가중치를 부여한다. (거리가 작을수록 가중치가 커진다)

위 그림에서 "d"를 확인하면, pixel-wise loss weight를 통해 경계를 더욱 명확하게 구분하고 있음을 확인할 수 있다.
3.1. Data Augmentation
Biomedical Image는 그 특성상 이미지의 개수가 많지가 않아서 data augmentation을 진행했다.
U-Net 논문에서는 추가적으로 "elastic deformation" 변형을 적용했다.
Elastic Deformation은 실제 deformation에 대해서도 invariance(불변성)을 학습할 수 있도록 한다.
Deformation은 tissue에서 발생하는 가장 흔한 변화 중 하나이고, 실제 deformation된 모습에 대해서도 사실적인 simulation이 가능하기 때문에 사용하였다.
* 참조 : https://www.nature.com/articles/srep27238

Augmentation을 통해 이를 구현함으로써 따로 deformation된 image들에 대한 별도의 라벨링 작업 없이 학습이 가능하다.
구체적인 방법은 다음과 같다. (잘 몰라서 따로 찾아봐야할 것 같다)
* 참조 : https://stat-cbc.tistory.com/28
1. 수평 및 수직 방향에 대한 가우스 분포를 따르는 임의의 stress(강도)가 생성
2. 가까운 픽셀이 유사한 변위(displacement)를 갖도록하기 위해 결과 수평 및 수직 이미지에 Gaussian 필터를 별도로 적용


논문에서 진행한 Experiment & 모델들과의 비교는 생략!!
끝!!
Reference
- https://ganghee-lee.tistory.com/44
- https://jlog1016.tistory.com/85
- https://www.youtube.com/watch?v=n_FDGMr4MxE
- https://medium.com/@msmapark2/u-net-%EB%85%BC%EB%AC%B8-%EB%A6%AC%EB%B7%B0-u-net-convolutional-networks-for-biomedical-image-segmentation-456d6901b28a
- https://velog.io/@minkyu4506/%EB%85%BC%EB%AC%B8-%EB%A6%AC%EB%B7%B0-U-Net-%EB%A6%AC%EB%B7%B0





댓글 영역